Páginas
▼
martes, 25 de diciembre de 2018
viernes, 21 de diciembre de 2018
El mono guarrete
El concepto caca, pedo, culo, pis sigue sirviendo de inspiración para crear juego estúpidos
Os juro que estoy esperando el artículo científico que explique porqué no nos molestan nuestros pedos y si los de los demás
Os juro que estoy esperando el artículo científico que explique porqué no nos molestan nuestros pedos y si los de los demás
lunes, 17 de diciembre de 2018
Destapa y atrapa
Tienen que pillar una mierda
al vuelo y competir con los demás para cogerla... Sólo 20 euros. Un juego educativo,
les prepara para su vida adulta, cuando tengan que aceptar cualquier
trabajo de mierda antes de que lo haga otro.
viernes, 14 de diciembre de 2018
jueves, 22 de noviembre de 2018
Materia fecal en los ríos significa brotes de diarrea
En estos momentos hay una alerta por E. coli 0157 en los EEUU en la variedad romana de lechugas ¿El problema? que se riegan con aguas contaminadas de heces de ganado y claro, como la lechuga no se cocina cuando tomamos estas ensaladas nos provocan una diarrea de las fuertes.
artículo que no te informa de nada.
Un virus convirtió a la inocente E. coli en un patógeno que causa diarrea hemorrágica
La bacteria E. coli es una bacteria que normalmente vive sin causar problemas en los intestinos de mamíferos de sangre caliente. Algunas de estas bacterias, como la variedad 0157.H7, han sido colonizadas por virus que les confieren la capacidad de provocar fuertes diarreas ¿Por qué? porque eso aumenta la transmisión de E. coli entre personas.
E. coli, cuando no está colonizada por el virus, es una bacteria que vive comensal en los humanos, vacas. Aunque la eliminamos con las heces como es de rápido crecimiento siempre habrá E. coli vivendo en nuestros intestinos. Estas E. coli portadoras de virus en su interior, como la 0157, tienen un comportamiento más vírico que bacteriano. Lo que necesita el virus es transmitirse, transmitirse y transmitirse entre hospedadores. Por ese motivo, entre los genes del virus están aquellos que hacen que los intestinos empiecen a secretar agua y nos obligue a estar defecando todo el rato. El virus de E. coli consigue que nos convirtamos en un aspersor, o en un espray de bacterias. Cagamos constantemente, nos manchamos e infectamos todo lo que está a nuestro alrededor. Si infectamos a otras personas o a otros animales el virus habrá triunfado.
El agua contaminada matará cada vez a más gente pobre
Esta lógica vírica funciona porque existe superpoblación de humanos y vacas. Si la E. coli 0157 no tuviese fácil su transmisión esta bacteria cada vez sería menos frecuente. El problema es que, viviendo en los márgenes de los cursos de agua, cada vez hay más humanos y más ganadería. A medida que cada vez hay más humanos y los cursos de agua son más escasos este problema será cada vez más frecuente.
Cuando la industria detecte esta bacteria en sus productos estará tentada de derivar estos productos a países en donde no existan controles y pueda ser vendida sin que se les eche a ellos la culpa de estar propagando un brote de E. coli 0157. Quizás se pueda tratar en el futuro con fagos anti-E. coli 0157 para así prevenir la dispersión de estas bacterias patógenas, pero claro, este tipo de tratamientos sólo estarán disponibles en países ricos y para establecimientos caros como, por ejemplo, la cadena de productos orgánicos Whole Foods de los Estados Unidos.
artículo que no te informa de nada.
Un virus convirtió a la inocente E. coli en un patógeno que causa diarrea hemorrágica
La bacteria E. coli es una bacteria que normalmente vive sin causar problemas en los intestinos de mamíferos de sangre caliente. Algunas de estas bacterias, como la variedad 0157.H7, han sido colonizadas por virus que les confieren la capacidad de provocar fuertes diarreas ¿Por qué? porque eso aumenta la transmisión de E. coli entre personas.
E. coli, cuando no está colonizada por el virus, es una bacteria que vive comensal en los humanos, vacas. Aunque la eliminamos con las heces como es de rápido crecimiento siempre habrá E. coli vivendo en nuestros intestinos. Estas E. coli portadoras de virus en su interior, como la 0157, tienen un comportamiento más vírico que bacteriano. Lo que necesita el virus es transmitirse, transmitirse y transmitirse entre hospedadores. Por ese motivo, entre los genes del virus están aquellos que hacen que los intestinos empiecen a secretar agua y nos obligue a estar defecando todo el rato. El virus de E. coli consigue que nos convirtamos en un aspersor, o en un espray de bacterias. Cagamos constantemente, nos manchamos e infectamos todo lo que está a nuestro alrededor. Si infectamos a otras personas o a otros animales el virus habrá triunfado.
El agua contaminada matará cada vez a más gente pobre
Esta lógica vírica funciona porque existe superpoblación de humanos y vacas. Si la E. coli 0157 no tuviese fácil su transmisión esta bacteria cada vez sería menos frecuente. El problema es que, viviendo en los márgenes de los cursos de agua, cada vez hay más humanos y más ganadería. A medida que cada vez hay más humanos y los cursos de agua son más escasos este problema será cada vez más frecuente.
 |
| El río Yamuma en Delhi, India, contiene un 100% de materia fecal. Fuente |
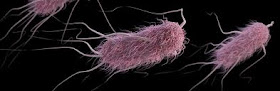 |
| Las Escherichia coli patógenas causan mucha alarma en los países ricos acostumbrados a no tener problemas con las bacterias. Por ese motivo, deberían llamarse Histeria coli |
miércoles, 21 de noviembre de 2018
Cómo la secuenciación masiva ha cambiado a la ciencia
Mi amiga Nuria Rodríguez ha grabado a científicos de Centro de Biología Molecular de Madrid sus opiniones sobre cómo la secuenciación masiva de ADN ha cambiado la ciencia
lunes, 19 de noviembre de 2018
Micropía
¿Se puede crear un relato museístico sobre las bacterias? en el Zoológico Artis de Amsterdam lo han hecho. Entre sus instalaciones se encuentra Micropía: una exhibición de microbios
En Holanda se sienten orgullosos de Leuwenhoek, la primera persona en descubrir para la humanidad el mundo de lo micro.
miércoles, 14 de noviembre de 2018
No aceptamos pacientes que no vacunen a sus hijos
“Nuestra clínica ya no atenderá a pacientes que no vacunen a sus hijos. Vacunaremos a aquellos que quieran comenzar el proceso, pero si se niegan a seguir con nuestra política, serán dados de alta a partir del 1 de Enero de 2019. Podemos proporcionar un volante de autorización cuando encuentren un nuevo médico. Debido a la abrumadora y meticulosa investigación científica y los descubrimientos basados en ella, hay una gran cantidad de pruebas que apoyan el impacto positivo de las vacunas en los niños. El consejo profesional del doctor Azmeh, junto con la CDC y la OMS, es desanimar a cualquier padre que no quiera vacunar a sus hijos, algo que sin duda les ayudará. Si tienen alguna duda o pregunta sobre esta política, por favor pregunten a nuestros empleados.”
lunes, 5 de noviembre de 2018
Disparates en los exámenes
Entiendes lo que es la labor docente cuando un alumno de 2º semestre contesta esto en un examen:
PREGUNTA: ¿Qué diferencias existen entre el ADN y el ARN en cuestión de bases nitrogenadas y azúcares? ¿Explican estas diferencias la estabilidad del ADN como molécula soporte de la herencia en la mayor parte de los seres vivos?
Respuesta que no dice nada: En este contexto las diferencias de bases y azúcares no son suficientes para explicar la estabilidad del ADN en la herencia pero si se sabe que esta lleva una base fundamental de todo ser.
PREGUNTA: Pedro Leiva notó mejoría de sus fiebres tercianas (malaria) cuando se bañó en un riachuelo de Malacatos (Loja) ¿Cómo aplicó el método científico para lograr su descubrimiento de la quina?
Respuestas:
PREGUNTA: ¿Qué diferencias existen entre el ADN y el ARN en cuestión de bases nitrogenadas y azúcares? ¿Explican estas diferencias la estabilidad del ADN como molécula soporte de la herencia en la mayor parte de los seres vivos?
Respuesta que no dice nada: En este contexto las diferencias de bases y azúcares no son suficientes para explicar la estabilidad del ADN en la herencia pero si se sabe que esta lleva una base fundamental de todo ser.
PREGUNTA: Pedro Leiva notó mejoría de sus fiebres tercianas (malaria) cuando se bañó en un riachuelo de Malacatos (Loja) ¿Cómo aplicó el método científico para lograr su descubrimiento de la quina?
Respuestas:
Otras más (aquí se ha logrado un record: seis verbos en una frase)
PREGUNTA: ¿Qué fue antes el huevo o la gallina?
Respuesta: Primero fue el huevo; porque la gallina tuvo un ancestro que fue quien puso el huevo. Este huevo.
PREGUNTA: ¿Qué podríamos hacer si la concentración de la proteína problema que queremos cuantificar es más concentrada que las concentraciones que hemos utilizado de BSA para realizar la curva de calibrado?
La respuesta correcta sería: diluír la muestra
Respuesta "Cantinflas molecular": Se hace una recombinación que establece la diferencia de las características de la solución
PREGUNTA: ¿Qué fue antes el huevo o la gallina?
Respuesta: Primero fue el huevo; porque la gallina tuvo un ancestro que fue quien puso el huevo. Este huevo.
PREGUNTA: ¿Qué podríamos hacer si la concentración de la proteína problema que queremos cuantificar es más concentrada que las concentraciones que hemos utilizado de BSA para realizar la curva de calibrado?
La respuesta correcta sería: diluír la muestra
Respuesta "Cantinflas molecular": Se hace una recombinación que establece la diferencia de las características de la solución
jueves, 25 de octubre de 2018
¿Por qué las arqueas no causan enfermedades?
Las membranas de las arqueobacterias no tienen, como en las eubacterias, pared celular de peptidoglicano. Son membranas monocapa. En su membrana, al igual que en las eucariotas, existen esteroles. Poseen lípidos de membrana distintos a los de las eubacterias y eucariotas: diéteres de glicerina con terpenos.
 |
| De la bacteria primordial de la que procede toda la vida, llamada LUCA, surgen dos grandes grupos, el de las eubacterias y el de las arqueobacterias. Cuando una arquea y una eubacteria, por endosimbiosis, generan la primera célula eucariota (con nucleo) se forma un nuevo grupo: eucaria formado por protozoos unicelulares y los pluricelulares conocidos como hongos, plantas y animales. De Maulucioni - Trabajo propio, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=33251850 |
Las eubacterias y las arqueas se parecen. Ambos tipos de organismos son unicelulares, carecen de nucleos y de organelos. Las arqueas tienen un tipo de membrana plasmática único. Cuando se examina su ARN ribosomal se observa que son un grupo único diferenciado de las eubacterias.
De cientos de microbios que causan enfermedades en humanos ninguno es arquea
En un artículo publicado en Bioessays, Erin Gill and Fiona Brinkman tratan de explicar el porqué. La hipótesis más trivial es la estadística, es decir, si sólo el 0.36% de las bacterias causan enfermedades en los humanos, es decir, de 151000 bacterias conocidas 585 son patógenos humanos, tirando por lo bajo, teniendo en cuenta especies que se pueden cultivar en laboratorio y las que no se pueden cultivar. Asumiendo que existe un número similar de especies de arqueobacterias patógenas en el grupo, si se conocen 4508 especies de arqueobacterias x 0.0036 = deberían haber 16 patógenos. Sin embargo, no existen 16 patógenos arqueobacterianos. Obviamente para llegar a esta conclusión debemos asumir que el porcentaje de patógenos en ambos grupos es el mismo lo cual es una asumción.
Recientemente, se ha estudiado la metagenómica (secuencias fragmentadas de ADN) de arqueas mesofílicas marinas. Podría haber, por tanto, muchísimas arqueas todavía no descubiertas y por tanto habría muchas enfermedades que todavía no han sido descritas. La microbiología nació como una especialidad médica y se ocupó básicamente de eubacterias que causaban enfermedades en los humanos. El grupo de las arqueobacterias, bacterias que viven en condiciones extremas para los humanos, fue estudiado por microbiólogos ambientalistas que las estudiaban para entender los ciclos biogeoquímicos y la diversidad de la vida, eran investigadores que no buscaban patógenos
Los bacteriófagos matan a las personas, las bacterias no tienen la culpa
Una pista acerca de cómo una bacteria se vuelve patógena podría estar en las islas de patogenicidad. Los genes de virulencia están ordenados en regiones específicas de las bacterias patógenas. Son regiones de origen vírico (plásmidos, transposones o bacteriófagos) que codifican proteínas que le permiten a la bacteria invadir nuestro cuerpo, evitar al sistema inmune y transmitirse de persona a persona.
Muchos de estos genes de virulencia son móbiles y pueden saltar del genoma de una bacteria a otra convirtiendo a la nueva bacteria en una bacteria patógena. El vector que lleva esos genes es frecuentemente un virus bacteriano llamado bacteriófago (fago). Es lo que se llama transducción. Brevemente, cuando un fago invade una bacteria genera muchas copias de si mismo, en ese proceso puede tomar genes de la bacteria invadida. Esos genes pasan a formar parte del genoma del fago que puede invadir otra especie. Si el fago inserta su genoma dentro del genoma de la nueva bacteria, ésta adquirirá los nuevos genes, convirtiéndose en una bacteria patógena. Es lo que se llama transferencia horizontal de genes en contraposición con la transmisión vertical de genes que es lo que se transmite de padres a hijos.
Las bacterias patogénicas son vehículos para que los fagos se transmitan
Por este motivo, podríamos ver a las bacterias como el ambiente en el que se replican, viven y se transmiten los bacteriófagos. Esto tiene una explicación. Cuando no existían bacterias, los virus eran las únicas entidades biológicas en la tierra. Eran unas células primitivas que en vez de tener una membrana tenían una cápside proteica. Para hacer copias de si mismos tenían que desarmar esa cápside y copiar su ADN. Podían hacer eso porque en ese tiempo el mar y el agua dulce terraqueo era una sopa biológica rica en nutrientes. En ese momento los virus eran un poco distintos a los de hoy en día. Tenían como molécula soporte de la herencia el ARN o el ADN, y para traducir tenían ribosomas.
LUCA, la primera célula, una procariota, fue tan eficiente metabólicamente que acabó con toda la sopa biológica. Por decirlo de alguna forma, la sopa biológica que estaba libre en los mares y cursos de agua ahora estaba dentro de la membrana de las bacterias. Por ese motivo, los virus tuvieron que ir donde estaba la sopa biológica: en el citoplasma de las bacterias.
De lo que se conoce hasta la fecha, los bacteriófagos, virus especializados en replicarse en eubacterias, no invaden arquea. Arquea tiene sus propios virus. La razón: los receptores a los que se unen los fagos no están presentes en las arqueas. Hay otra razón, las Gram negativas tienen una presión interna de 5 atmósferas, las Gram positivas de 25 atmósferas. Por ese motivo, los fagos son una mezcla de virus unidos a una bomba neumática, para inyectar ADN contra la presión interna de la bacteria, y unas patas como de mosquito. Las arqueas no tiene presión interna, por eso no hace falta inyectar a presión el ADN.
Cuando comparamos los genomas de los virus que invaden arqueas con el de los fagos vemos que son bastante distintos. Los fagos no invaden arqueas y los virus de arqueas no invaden eubacterias.
 |
Arqueas tienen una membrana plasmática distinta a las de las bacterias y carecen de proteínas receptoras para los fagos por eso tienen sus propios virus. En la imagen la arquea Sulfolobus infectada por el virus STSV1. Fuente |
Para saber más:
paper published in Bioessays,
http://bytesizebio.net/2011/03/16/why-are-there-no-disease-causing-archaea/
martes, 23 de octubre de 2018
No pueden aceptar un camino sin salida
El siglo pasado, los físicos crearon la mecánica cuántica [su modelo matemático] para explicar cómo se comportan las cosas muy pequeñas, átomos y electrones, mientras que Einstein produjo su teoría de la relatividad general para explicar el comportamiento de objetos enormes como las galaxias.
Ambas teorías funcionan bien, pero son incompatibles. La física cuántica no puede explicar las cosas muy masivas y la relatividad general no puede aplicarse a lo muy pequeño. En comparación, los biólogos tienen en este sentido a la teoría de la selección natural de Darwin para explicar los seres vivos, grandes y pequeños, desde las ballenas hasta las bacterias. Sin embargo, como decimos, los físicos no tienen un código unificado. Una perspectiva que molestó tanto por ejemplo al mismísimo Einstein que se pasó los últimos 20 años de su vida buscando infructuosamente una teoría que lo pudiese unificar todo. Fuente
Una parte del problema, dicen los críticos, es que en los años ochenta los profesores alentaron a casi todos los jóvenes físicos con talento a estudiar la teoría de cuerdas debido a su inmensa promesa. Ahora son jefes de departamento de mediana edad que han comprometido sus vidas a dicho modelo y que no pueden aceptar a estas alturas que todo sea un camino sin salida.
domingo, 21 de octubre de 2018
Protozoo (unicelular) se come a un rotífero (pluricelular)
Video de un Loxophylum meleagris, un organismo unicelular gigante,
comiéndose a un rotífero, un organismo compuesto por más de 1000 células
viernes, 19 de octubre de 2018
martes, 16 de octubre de 2018
Los Nobel premian la evolución
El premio Nobel de Química del 2018 ha sido para los investigadores que han utilizado la evolución molecular para mejorar proteínas ya existentes. George Smith y Greg Winter son dos de los ganadores de la mitad del Nobel de Química del 2018 porque la otra mitad de este Nobel ha sido para Frances Arnold.
Los fagos muestran el camino de la proteína al gen
Smith en 1985 empezó a trabajar con bacteriófagos (fagos) para poder clonar genes. Sin embargo, casi por casualidad, descubrió que podía a partir de una proteína descubrir el gen que la codificaba. Es como si por ejemplo, tuviésemos una fotografía y pudiésemos localizar en el disco duro el código máquina, de ceros y unos que la codifica.
Los fagos son virus que se replican en las bacterias. Como las bacterias son pequeñas células a presión, los fagos son virus que tienen una especie de bomba de bicicleta acoplada con la que pueden inyectar el ADN (o el ARN) en el interior de la célula. En los ochenta había muchas bibliotecas de fragmentos de ADN en fagos. Con la tecnología de aquel entonces se podía hacer corta pega de fragmentos de ADN humano, por ejemplo, en el interior del genoma de un fago. De esta manera, podíamos tener la oportunidad que la proteína se expresase en el exterior del fago.
Ahora que teníamos la proteína humana expresada en la superficie del fago había que pescarla.
Anticuerpos como anzuelos
Ahora que teníamos muchísimos fagos portando distintas proteínas en su superficie, lo siguiente era utilizar anticuerpos para pescarlos. Y esto se podía hacer en la década de los ochenta. Inyectas una proteína pura en un conejo y el sistema inmune del animal fabrica anticuerpos contra esa proteína. De esta forma podían unir fagos que expresaban la proteína humana en su superficie a los anticuerpos. Y como el fago adherido en el anticuerpo, en su interior, llevaba el gen, el código, podían saber qué gen era el responsable de producir esa proteína humana en cuestión: encontrar una aguja en un pajar de manera elegante.
Greg Winter llevó esta técnica a otro nivel...
Anticuerpos que bloquean el desarrollo de enfermedades
El sistema inmune humano puede producir cientos de miles de anticuerpos diferentes. Las células del sistema inmune humano que producen anticuerpos que reaccionan contra moléculas humanas son destruídos (cuando no se destruyen todas es cuando tenemos enfermedades autoinmunes, es decir, que reaccionan contra nosotros mismos). Sin embargo, las células del sistema inmune que no reaccionan contra nosotros mismos patrullan por los vasos sanguíneos. Cuando una de ellas reconoce una bacteria o un virus comienza a multiplicarse. Por ese motivo son tan eficaces eliminando patógenos extraños del cuerpo humano.
Los anticuerpos son muy selectivos. Son capaces de reconocer una molécula entre decenas de miles de otras moléculas ¿Es posible seleccionar anticuerpos que se unan a moléculas propias de enfermedades humanas y las bloqueen y así detengan a la enfermedad?
Winter puso anticuerpos en la superficie de los fagos
Los anticuerpos son proteínas que tienen forma de Y. Así que Winter fue capaz de pegar esta proteína a la superficie de un fago. De forma que la información, el código de ADN, para formar esa proteína estuviese en el genoma del fago. El usó un anticuerpo que se unía a una pequeña molécula conocida como phOx por que se conocía la secuencia genética de este anticuerpo, por eso fue capaz de meter la secuencia en el genoma de un fago y así este fago llevaba el anticuerpo anti-phOx en su genoma.
De esta manera, Winter fue capaz de recuperar el fago expresando el anticuerpo anti-phOx, poniendo phOx en una sopa de 4 millones de fagos, uno de los cuales era el anti-phOx. Lo que hizo con este sistema fue repetir este buscar una aguja en un pajar varias veces. Lo que observó es que a cada ciclo la unión del anticuerpo producido por el fago a phOx era cada vez más fuerte. Esto señores y señoras no es otra cosa que evolución por selección natural. En 1994 utilizando este método fue capaz de seleccionar anticuerpos con una altísima afinidad a células cancerígenas.
Se crea la primera farmaceutica basada en anticuerpos humanos
La compañía se llama adalimumab. Esta compañía creo anticuerpos que neutralizaban la proteína TNF-alfa. Inactivando esta proteína se detiene la inflamación asociada a varias enfermedades autoinmunes. En 2002 se aprobaron anticuerpos contra la artritis reumatoide, la psoriasis y la enfermedad inflamatoria intestinal. Otra aplicación ha sido crear anticuerpos que neutralizan la toxina del antrax. En este momento se trabaja en anticuerpos contra el Alzheimer y contra el lupus.
Selección artificial, lo mismo que hacen los criadores de perros
Lo bonito de esta idea es su simplicidad. Para lograr un anticuerpo que se una a su diana de manera superfuerte no hace falta saber de química orgánica... lo único que hace falta es que los virus creen trillones de copias y que esas copias tengan errores. Alguno de esos errores, mutaciones, van a dar un anticuerpo mejorado. Ese anticuerpo se selecciona y se le deja que vuelva a dar millones de copias, todas parecidas a él menos unos miles de mutantes. Si alguno de esos mutantes se adhiere con más fuerza, se le deja que vuelva a producir millones de copias. Alguno habrá que, al haber mutado, tenga una mejora que lo haga más afín y más fuerte.
Es lo que ha hecho la humanidad desde el neolítico en que las mujeres aprendieron a seleccionar semillas y animales domésticos. Todas las razas de perros proceden de los lobos. Cuando se mataban a los lobos adultos alguna mujer a la que su hijo se había destetado cogía un cachorro y lo alimentaba con su propio pecho. Al margen de que los humanos sentimos ternura innata por los cachorrillos, esto le servía a la mujer porque sabían que las mujeres lactantes tienen más dificultades para quedarse de nuevo embarazada. Cuando el cachorro crecía seguía teniendo un vínculo emocional con su madre adoptiva. Los cachorros adultos tienen sobre 8 cachorritos todos los años. No se los puede mantener a todos, por lo que se matan en su mayoría. Solo se les deja vivir a aquellos cachorros que tienen una característica que nos interesa. Si vivimos en una zona de frío que sean peludos, si es una zona calientes con poco pelo. Si los queremos para comer los escogemos pequeños, si los queremos para que nos defiendan dejamos los musculosos.
Scientific Background
Audio programa Efervesciencia (en galego):
Los fagos muestran el camino de la proteína al gen
Smith en 1985 empezó a trabajar con bacteriófagos (fagos) para poder clonar genes. Sin embargo, casi por casualidad, descubrió que podía a partir de una proteína descubrir el gen que la codificaba. Es como si por ejemplo, tuviésemos una fotografía y pudiésemos localizar en el disco duro el código máquina, de ceros y unos que la codifica.
Los fagos son virus que se replican en las bacterias. Como las bacterias son pequeñas células a presión, los fagos son virus que tienen una especie de bomba de bicicleta acoplada con la que pueden inyectar el ADN (o el ARN) en el interior de la célula. En los ochenta había muchas bibliotecas de fragmentos de ADN en fagos. Con la tecnología de aquel entonces se podía hacer corta pega de fragmentos de ADN humano, por ejemplo, en el interior del genoma de un fago. De esta manera, podíamos tener la oportunidad que la proteína se expresase en el exterior del fago.
Ahora que teníamos la proteína humana expresada en la superficie del fago había que pescarla.
Anticuerpos como anzuelos
Ahora que teníamos muchísimos fagos portando distintas proteínas en su superficie, lo siguiente era utilizar anticuerpos para pescarlos. Y esto se podía hacer en la década de los ochenta. Inyectas una proteína pura en un conejo y el sistema inmune del animal fabrica anticuerpos contra esa proteína. De esta forma podían unir fagos que expresaban la proteína humana en su superficie a los anticuerpos. Y como el fago adherido en el anticuerpo, en su interior, llevaba el gen, el código, podían saber qué gen era el responsable de producir esa proteína humana en cuestión: encontrar una aguja en un pajar de manera elegante.
Greg Winter llevó esta técnica a otro nivel...
Anticuerpos que bloquean el desarrollo de enfermedades
El sistema inmune humano puede producir cientos de miles de anticuerpos diferentes. Las células del sistema inmune humano que producen anticuerpos que reaccionan contra moléculas humanas son destruídos (cuando no se destruyen todas es cuando tenemos enfermedades autoinmunes, es decir, que reaccionan contra nosotros mismos). Sin embargo, las células del sistema inmune que no reaccionan contra nosotros mismos patrullan por los vasos sanguíneos. Cuando una de ellas reconoce una bacteria o un virus comienza a multiplicarse. Por ese motivo son tan eficaces eliminando patógenos extraños del cuerpo humano.
Los anticuerpos son muy selectivos. Son capaces de reconocer una molécula entre decenas de miles de otras moléculas ¿Es posible seleccionar anticuerpos que se unan a moléculas propias de enfermedades humanas y las bloqueen y así detengan a la enfermedad?
Winter puso anticuerpos en la superficie de los fagos
Los anticuerpos son proteínas que tienen forma de Y. Así que Winter fue capaz de pegar esta proteína a la superficie de un fago. De forma que la información, el código de ADN, para formar esa proteína estuviese en el genoma del fago. El usó un anticuerpo que se unía a una pequeña molécula conocida como phOx por que se conocía la secuencia genética de este anticuerpo, por eso fue capaz de meter la secuencia en el genoma de un fago y así este fago llevaba el anticuerpo anti-phOx en su genoma.
De esta manera, Winter fue capaz de recuperar el fago expresando el anticuerpo anti-phOx, poniendo phOx en una sopa de 4 millones de fagos, uno de los cuales era el anti-phOx. Lo que hizo con este sistema fue repetir este buscar una aguja en un pajar varias veces. Lo que observó es que a cada ciclo la unión del anticuerpo producido por el fago a phOx era cada vez más fuerte. Esto señores y señoras no es otra cosa que evolución por selección natural. En 1994 utilizando este método fue capaz de seleccionar anticuerpos con una altísima afinidad a células cancerígenas.
Se crea la primera farmaceutica basada en anticuerpos humanos
La compañía se llama adalimumab. Esta compañía creo anticuerpos que neutralizaban la proteína TNF-alfa. Inactivando esta proteína se detiene la inflamación asociada a varias enfermedades autoinmunes. En 2002 se aprobaron anticuerpos contra la artritis reumatoide, la psoriasis y la enfermedad inflamatoria intestinal. Otra aplicación ha sido crear anticuerpos que neutralizan la toxina del antrax. En este momento se trabaja en anticuerpos contra el Alzheimer y contra el lupus.
Selección artificial, lo mismo que hacen los criadores de perros
Lo bonito de esta idea es su simplicidad. Para lograr un anticuerpo que se una a su diana de manera superfuerte no hace falta saber de química orgánica... lo único que hace falta es que los virus creen trillones de copias y que esas copias tengan errores. Alguno de esos errores, mutaciones, van a dar un anticuerpo mejorado. Ese anticuerpo se selecciona y se le deja que vuelva a dar millones de copias, todas parecidas a él menos unos miles de mutantes. Si alguno de esos mutantes se adhiere con más fuerza, se le deja que vuelva a producir millones de copias. Alguno habrá que, al haber mutado, tenga una mejora que lo haga más afín y más fuerte.
Es lo que ha hecho la humanidad desde el neolítico en que las mujeres aprendieron a seleccionar semillas y animales domésticos. Todas las razas de perros proceden de los lobos. Cuando se mataban a los lobos adultos alguna mujer a la que su hijo se había destetado cogía un cachorro y lo alimentaba con su propio pecho. Al margen de que los humanos sentimos ternura innata por los cachorrillos, esto le servía a la mujer porque sabían que las mujeres lactantes tienen más dificultades para quedarse de nuevo embarazada. Cuando el cachorro crecía seguía teniendo un vínculo emocional con su madre adoptiva. Los cachorros adultos tienen sobre 8 cachorritos todos los años. No se los puede mantener a todos, por lo que se matan en su mayoría. Solo se les deja vivir a aquellos cachorros que tienen una característica que nos interesa. Si vivimos en una zona de frío que sean peludos, si es una zona calientes con poco pelo. Si los queremos para comer los escogemos pequeños, si los queremos para que nos defiendan dejamos los musculosos.
 |
| Los mismo que en el neolítico seleccionamos razas de perros a partir del lobo, hoy en día estamos aprendiendo a domesticar y seleccionar las moléculas y de nuevo, la selección es la herramienta. |
Audio programa Efervesciencia (en galego):
Detectar para eliminar
 |
| Fleming observó, al poco tiempo del uso de la penicilina, que había bacterias capaces de resistir al flamante nuevo antibiótico. |
 |
| La penicilina curaba la gonorrea en 4 horas en los años 40 del siglo pasado. Hoy en día estamos a punto de que aparezcan bacterias de la gonorrea resistentes a todos los antibióticos conocidos |
 |
El negocio pollos-antibióticos está tan ligado que existen compañías, como Venky en la India, que se dedican a la producción de pollos y de antibióticos. |
 |
| Eugenio Espejo, uno de los padres de la teoría microbiana de la enfermedad, propuso la creación de una policía higiénica. Una medida muy necesaria hoy en día. |
Las personas nos encontramos indefensas frente a la dinámica de maximización de beneficios de las empresas. Si usar antibióticos es bueno para lograr esos beneficios va a ser muy difícil detenerlas. Por ahora, solo nos queda utilizar la higiene, es decir, establecer una barrera para la difusión de las bacterias resistentes a los antibióticos allí donde hay más probabilidad de que aparezcan: las granjas, especialmente las granjas de pollos.
¿Cómo vigilaremos que las bacterias con resistencia a antibióticos de pollos no lleguen a los consumidores?
Para empezar tendremos que vigilar aquellos genes que se pueden transmitir de una bacteria a otra. Son genes que producen betalactamasas o carbapenemasas capaces de destruir a los antibióticos betalactámicos o los carbapenémicos. Esos genes se encuentran muchas veces en plásmidos, que son moléculas de ADN que se pueden intercambiar entre bacterias. Esto lo hace muy peligroso porque si las bacterias están muy juntas, y en las granjas y en las balsas de lixiviados lo están, se pueden intercambiar genes como los niños intercambian cromos.
Si detectamos qué bacterias son las portadoras de estos genes que destruyen los antibióticos y que estos genes están en elementos móviles que pueden pasar a nuestras bacterias, entonces ya tendremos frente a nosotros a la bacteria que debemos controlar y en el futuro, tratar de eliminar.
jueves, 11 de octubre de 2018
Barreras para la transmisión de coliformes multirresistentes
Introducción: Los filtros cerámicos (FC) para el tratamiento domiciliar del agua (TDA) son una solución económica para poblaciones rurales dispersas que, en Ecuador, carecen en un 60%, de agua potable. En este trabajo examinamos la carga bacteriana y su resistencia a antibióticos y la capacidad de los FC para evitar la dispersión de bacterias resistentes.
Métodos: Se distribuyeron FC a familias sin acceso a agua potable con hijos menores de 15 años de Guanábana (17 familias ) y Vista Hermosa (20 familias) en la parroquia de Gualea, Quito, Ecuador. Se analizaron 100 ml del agua de beber, antes y meses después del empleo del FC. Las bacterias presentes se aislaron en filtros de 0.45 um de diámetro y se crecieron en placas con el medio de cultivo cromogénico Chromo Cult. Las colonias de coliformes y Escherichia coli (E. coli) se cuantificaron y se crecieron en medio líquido con ceftriaxona (3ug/mL). Se analizó su perfil de resistencia antibiótica mediante el sistema Phoenix.
Resultados: Antes del empleo del FC se obtuvo una media de UFC/100 ml por familia de 41 E. coli y 2421 de coliformes en Guanábana. En Vista Hermosa se obtuvo 34 de E. coli y 1843 de coliformes. En 17 muestras se aislaron E. coli con resistencia a cefalosporinas de tercera genaración mediada por Beta-lactamasas de Espectro Extendido (BLEE) y resistencia a ≥3 familias de antimicrobianos (multiresistentes). No se detectó resistencia a ertapemen o colistina. Después de emplear FC en Guanábana se obtuvo 0 de E. coli y 30 de coliformes y en Vista Hermosa 0 de E. coli y 21 de coliformes. Ninguna de estas colonias presentó resistencia a antibióticos.
Conclusiones: Estos resultados sugieren que los FC para TDA son efectivos para evitar la dispersión de bacterias resistentes a antibióticos en poblaciones rurales dispersas sin agua potable.
Agradecemos al programa de Vinculación de la UDLA, en especial a su directora Luz Dary Ulloa, por el apoyo a este proyecto.
Métodos: Se distribuyeron FC a familias sin acceso a agua potable con hijos menores de 15 años de Guanábana (17 familias ) y Vista Hermosa (20 familias) en la parroquia de Gualea, Quito, Ecuador. Se analizaron 100 ml del agua de beber, antes y meses después del empleo del FC. Las bacterias presentes se aislaron en filtros de 0.45 um de diámetro y se crecieron en placas con el medio de cultivo cromogénico Chromo Cult. Las colonias de coliformes y Escherichia coli (E. coli) se cuantificaron y se crecieron en medio líquido con ceftriaxona (3ug/mL). Se analizó su perfil de resistencia antibiótica mediante el sistema Phoenix.
Conclusiones: Estos resultados sugieren que los FC para TDA son efectivos para evitar la dispersión de bacterias resistentes a antibióticos en poblaciones rurales dispersas sin agua potable.
Agradecemos al programa de Vinculación de la UDLA, en especial a su directora Luz Dary Ulloa, por el apoyo a este proyecto.
jueves, 27 de septiembre de 2018
Ejercicios crecimiento microbiano
 |
| Las bacterias crecen de manera exponencial, de una pasan a dos, de dos a cuatro, de cuatro a ocho y así sucesivamente. |
Log 2 (1024) = 10
¿Cómo cambiar la base del logaritmo en la calculadora?
En la calculadora científica existen dos teclas para resolver logaritmos:
La tecla «log» que sirve para resolver logaritmos de base 10
La tecla «ln» que sirve para calcular los logaritmos neperianos (en base e)
Es decir, directamente con la calculadora, sólo se pueden resolver logaritmos en base 10 o logaritmos neperianos. No podemos cambiar la base del logaritmo en la calculadora.
Por eso, si necesitamos resolver un logaritmo de cualquier otra base, necesitamos aplicar la fórmula del cambio de base.
¿Y qué es la fórmula del cambio de base?
La fórmula del cambio de base nos permite calcular logaritmos de cualquier base con la calculadora. Es la siguiente:
Lo que viene a decir esta fórmula es que si
tenemos el logaritmo en base «a» de un número N, podemos elegir
otra base «b», que puedes ser la que queramos y dividir el
logaritmo en esa base «b» del número entre el logaritmo en esa
base del número «a» («a» es la base del logaritmo original).
Fórmula del cambio de base con logaritmos en base 10
Como podemos elegir la base «b» que queramos,
vamos a jugar con eso a nuestro favor y vamos a elegir una base que
nos convenga, como es la base 10, que es la que tenemos en la tecla
«log» de la calculadora para resolver logaritmos.
Eligiendo la base 10, la fórmula del cambio de
base queda mucho más simplificada:
Ahora, el logaritmo en base «a» de un número N
es igual al logaritmo de N entre el logaritmo de «a» (ambos en base
10. Al estar en base 10, ya podemos resolverlos con la calculadora y
la fórmula se convierte en una simple división.
Vamos a verlo con un ejemplo: Resolver el
siguiente logaritmo:
Tenemos que resolver el logaritmo en base 7 de
100.
Aplicamos la fórmula del cambio de base y nos
queda que ese logaritmo, «log N» es el logaritmo de 100 y «log a»
es el logaritmo de 7, que es la base del logaritmo original.
Hallamos el valor de cada uno con la calculadora y
realizamos la división, obteniendo el valor del logaritmo original:
¡Funciona!
Tienes que sólo tienes que dividir el logaritmo
del número entre el logaritmo de la base del logaritmo original.
Existe una alternativa y es descargar Photomath en tu celular. Se escanea la fórmula y ¡Ya!
Fórmula del cambio de base con logaritmos neperianos
Como tenemos la tecla «ln», que también resuelve logaritmos, podemos utilizar la fórmula del cambio de base eligiendo la base e, es decir, los logaritmos neperianos, quedando de la siguiente forma:
El logaritmo en cualquier base de un número es el logaritmo neperiano del número entre el logaritmo neperiano de la base del logaritmo original.
Vamos a ver otro ejemplo resolviendo un logaritmo con la fórmula del cambio de base con logaritmos neperianos. Tenemos que resolver el logaritmo en base 5 de 123. El logaritmo en base 5 de 123 es igual al logaritmo neperiano de 123 entre el logaritmo neperiano de 5. Estos logaritmos neperianos ya los podemos resolver con la calculadora. Resultado 2.98.
Ejercicios:
1. i) Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 10.000 genes y se divide en dos bacterias cada 40 minutos. La bacteria B tiene 5000 genes. ¿Cuánto tiempo tarda B en dividirse?. ii) ¿Cuántas bacterias dejará de descendencia la bacteria A y cuántas la bacteria B tras 4 horas de crecimiento?
2. Clostridium difficile se divide cada 12 minutos. ¿Cuánto tiempo toma para que esta bacteria alcance 9,223372037×10¹⁸ descendientes?
3. En el problema del trigo y el ajedrez se muestra el poder de la progresión exponencial. Si nosotros incubamos una bacteria en presencia de antibiótico, como podemos ver abajo en el video, en la sección izquierda, la mayoría de las bacterias morirán en presencia de la penicilina. Si tenemos 9,223372037×10¹⁸ y se mueren el 99.99999% de las bacterias ¿Cuántas bacterias quedarían?
Las bacterias crecen en presencia de penicilina. Las de la izquierda son sensibles a la penicilina. Las bacterias de la derecha son resistentes al antibiótico
4. Comienzo un cultivo bacteriano a partir de 1 bacteria. Si se trata de Escherichia coli que se divide cada 20 minutos ¿Cuántas bacterias tendré a las 5 horas?
5. Si comienzo un cultivo 1 bacteria y al cabo de 6 horas tengo 16384 bacterias ¿Cuál será el tiempo medio de división de esta bacteria?
6. Después de un tratamiento antibiótico en el que han muerto el 99.99999% de las bacterias resulta que tengo 9,22x1011 bacterias. ¿Cuántas bacterias tenía en la infección antes de comenzar el tratamiento antibiótico?
7. i) Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 15000 genes y se divide en dos bacterias cada 40 minutos. La bacteria B tiene 5000 genes. ¿Cuánto tiempo tarda B en dividirse?. ii) ¿Cuántas bacterias dejará de descendencia la bacteria A y cuántas la bacteria B tras 5 horas de crecimiento?
8. i) Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 8000 genes y se divide en dos bacterias cada 40 minutos. La bacteria B tiene 5000 genes. ¿Cuánto tiempo tarda B en dividirse?. ii) ¿Cuántas bacterias dejará de descendencia la bacteria A y cuántas la bacteria B tras 3 horas de crecimiento?
9. Cuando no existían bacterias toda la vida sobre la Tierra era vírica. Eran protovirus de ARN que tenían ribosomas. Estos virus se replicaban en la sopa biológica (en Ecuador en el locro biológico). Cuando aparecieron las bacterias, éstas tenían una membrana que diferenciaba fuera de dentro. Las bacterias metieron dentro todo lo que les interesaba de la sopa biológica. Los protovirus al quedarse sin su alimento sufrieron una presión selectiva que favoreció a aquellos virus que sabían como penetrar en el interior de las bacterias y alimentarse de lo que antaño había sido suyo. Nacieron los virus como entidades parásitas de las células. Muchos biólogos siguen diciendo, de forma equivocada, que los virus no son entidades vivas porque se tienen que alimentar de una célula metabólicamente activa. Si es por eso, podríamos incluir en la categoría de virus a muchas personas que no son capaces de subsistir sin su tarjeta de crédito. ¿Cómo perdieron los protovirus sus ribosomas? porque hoy en día los virus actuales no tienen ribosomas. Como siempre la solución está en la selección natural. Imaginemos dos virus A y B. El virus A es un protovirus, tiene ribosomas, y su ARN tiene 60.000 bases. El virus B perdió los genes de los ribosomas y por esa razón tienen 40.000 bases. No necesita codificar ribosomas porque puede utilizar los de la bacteria que infecta. Ambos virus infectan y se replican en el interior de bacterias. Si la ARN polimerasa copia a una velocidad de 1000 bases por minuto, y en los cromosomas de A y B existe un solo ORI. ¿Cuánto tiempo tarda cada virus en replicarse? ¿Cuánta descendencia tendrá cada virus al cabo de seis horas?
10. Cuando entra un virus en una célula ¿Qué ribosoma utilizará para traducir su ARNm, un ribosoma viral o un ribosoma de la célula humana? ¿Por qué?
11. Imaginemos que una expedición a Marte se encuentra agua bajo una gruesa capa de polvo y allí encuentran bacterias marcianas. Se aseguran de que esas bacterias no proceden de una contaminación traída por la propia expedición. ¿Tendrían el mismo código genético que las bacterias de la Tierra? ¿Por qué?
12. Tenemos dos virus A y B. El virus A tiene una tasa de letalidad del 0.1%, similar al de la gripe estacional. El virus B tiene una tasa de letalidad del 10%. En epidemiología el número reproductivo básico R0 es el número promedio de casos nuevos que genera un caso dado a lo largo de un período infeccioso.
La R0 de A y B es de 5 en ambos Si ambos virus
infectan 1000 personas cada uno y esas personas vuelven a infectar
otras personas y éstas a otras, es decir, tres ciclos de
transmisión. Al final de esos dos ciclos de transmisión ¿Cuántas
han muerto? ¿Cuántas desarrollaron anticuerpos?
13. El 14 de mayo de 2020 en España se han registrado 27321 muertos por SARS-CoV-2. Al mismo tiempo un estudio estima que el 5% de los españoles (47 millones). Si se ha calculado que la tasa de letalidad del virus es 0.5-1%. ¿Tienen sentido estas cifras?
14. Si en España con 47 millones de habitantes se contagian con el SARS-CoV-2 el 60% de personas o el 80% ¿Cuántos muertos habría teniendo en cuenta que la tasa de letalidad es del 1%? ¿Y en Ecuador, con 17 millones de habitantes, cuántos muertos habría?
15. El genoma del SARS-CoV-2 (ARNm positivo) tiene alrededor de 30.000 nt. Observa el principio y el final de la secuencia del SARS-CoV-2. ¿Qué observas que te llame la atención? ¿Qué función tiene?
16. Respecto al crecimiento de las especies... a) ¿Qué gran diferencia existe entre los virus y los organismos basados en células? b) ¿Qué diferencia existe entre los seres unicelulares y los pluricelulares?
17. ¿Qué le ocurriría a un virus cuando por una mutación su R0 es menor que 1?
Un ratio, en definitiva, es la razón o cociente de dos magnitudes relacionadas entre sí. Siempre se divide la magnitud mayor por la menor. El valor tiene que ser positivo.
20. Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 10.000 genes y se divide en dos bacterias cada 40 minutos. La bacteria B tiene 5000 genes y se divide cada 20 min. Tras 5 horas de crecimiento ¿Cuántas más bacterias (ratio) dejará de descendencia la bacteria A respecto a B?
21. Tenemos dos virus A y B. El virus A tiene una tasa de letalidad del 10% y un R0 de 5. En esta cepa de virus ha aparecido un mutante, que llamaremos B, con una tasa de letalidad menor, de 0.1. Si A y B infectan cada uno 1000 personas, y estas personas infectan a otras personas, lo que llamaremos transmisión primera y las personas infectadas en la transmisión primera infectan a otras personas, que llamaremos transmisión segunda. ¿Cuántas personas acabarán siendo infectadas por A y por B? ¿Cuántas personas en estas dos transmisiones matarán el virus A y su mutante el virus B?
17. ¿Qué le ocurriría a un virus cuando por una mutación su R0 es menor que 1?
18. Un cultivo de bacterias parte de 106 individuos. Cada hora
se duplican. Cuando el cultivo alcanza los 9.450.000.000 individuos
a) ¿Cuántas horas han transcurrido? b) ¿Cuántas bacterias habrá
después de 12 hr? c) Si tenemos otra especie bacteriana que se
triplica cada hora y cuya población inicial era de 1.2 millones de bacterias
¿Cuál de los dos cultivos será más numeroso al cabo de 4 horas?
19.Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 10.000 genes y se divide en dos bacterias cada 50 minutos. La bacteria B tiene 5000 genes y se divide cada 25 min. Tras 6 horas de crecimiento ¿Cuántas más bacterias (ratio) dejará una bacteria respecto a la otra?
19.Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 10.000 genes y se divide en dos bacterias cada 50 minutos. La bacteria B tiene 5000 genes y se divide cada 25 min. Tras 6 horas de crecimiento ¿Cuántas más bacterias (ratio) dejará una bacteria respecto a la otra?
Un ratio, en definitiva, es la razón o cociente de dos magnitudes relacionadas entre sí. Siempre se divide la magnitud mayor por la menor. El valor tiene que ser positivo.
20. Tengo dos bacterias: A y B. La bacteria A tiene un cromosoma con 10.000 genes y se divide en dos bacterias cada 40 minutos. La bacteria B tiene 5000 genes y se divide cada 20 min. Tras 5 horas de crecimiento ¿Cuántas más bacterias (ratio) dejará de descendencia la bacteria A respecto a B?
21. Tenemos dos virus A y B. El virus A tiene una tasa de letalidad del 10% y un R0 de 5. En esta cepa de virus ha aparecido un mutante, que llamaremos B, con una tasa de letalidad menor, de 0.1. Si A y B infectan cada uno 1000 personas, y estas personas infectan a otras personas, lo que llamaremos transmisión primera y las personas infectadas en la transmisión primera infectan a otras personas, que llamaremos transmisión segunda. ¿Cuántas personas acabarán siendo infectadas por A y por B? ¿Cuántas personas en estas dos transmisiones matarán el virus A y su mutante el virus B?
Soluciones:
1.- i) 20 minutos ii) A 64 bacterias B 4096 bacterias
2.- Para resolver este problema utilizamos el logaritmo binario.
2y = x ; 2y = 9,223372037×10¹⁸
log29,223372037×10¹⁸ = y ; y = 63
En la calculadora del celular, o de la computadora se resuelve así
ln (n)
y = ------------
ln(2)
En donde y sería el resultato, es decir, 63 y n sería 9,223372037×10¹⁸
3.- 922337203700 bacterias, o sea 9,223372037x1011
100-99.99999 = 0.00001
0.00001 -------------- 100
x -------------- 9,22337203700x1018
x = 9,223372037x1011
4.- 32768 bacterias
5.- Ha habido 14 divisiones. Seis horas son 360 minutos. Divido 360 minutos entre las 14 divisiones y obtengo un tiempo medio de 25.7 minutos, o sea 26 minutos con 18 segundos.
6.- Si 9,22x1011-------- 0.00001%
x -------- 100%
x = 9,22x1011 x 100 / 0.00001 = 9,22x1018
7.- Bacteria A 181 bacterias; bacteria B 5.9x106
8.- Bacteria A: 22 bacterias después de 180 minutos. Bacteria B 147 bacterias.
9.- Virus A 30 minutos virus B 20 minutos. En 6 horas tendremos de A 4096 virus, y de B tendremos 262144. Si en vez de horas hablásemos de miles de años podemos entender porqué los virus actuales carecen de ribosomas.
10. Los virus no tienen ribosomas propios, utilizan siempre el ribosoma de la célula hospedadora. Los virus perdieron los ribosomas propios cuando aparecieron las primeras células porque aquellos virus que carecían de ellos se replicaban mucho más rápido, al tener menos genes, que los protovirus que si los poseían.
11. Tendrían códigos genéticos distintos porque carecerían de un antepasado común.
12. A infecta 1000 personas, una
muere y quedan 999 personas. Cuando infectan otras 5
personas 5x999=4995. Mueren cuatro
quedan 4991. Si
estas 4991 infectan cada una 5 personas tenemos 24955, si mueren 24
quedan 24931. En tres
transmisiones han muerto 29
personas.
B infecta 1000
personas, mueren 100 quedan 900.
Cuando infectan otras 5 personas 5x900=4500. Mueren 450
quedan 4050. Estas 4050x5= 20250, de estas personas se mueren
2025 quedan 18225. En
tres transmisiones han muerto 2575
personas
13. Si el 5% de los españoles son seropositivos para el SARS-CoV-2, es
decir, ya han pasado la infección. De ese 5% se han muerto 27321. 5%
de españoles son 2.350.000 personas. Si consideramos que la tasa de
letalidad es 0.5%. De 2.350.000 personas se habrían muerto 11.750
personas, cuando en realidad se han muerto 27321. Si la tasa de
letalidad fuese de 1%, de 2.350.000 personas (el 5% de los españoles)
los muertos serían 23,500.
Conclusión.
Si los muertos son 27321 puede pasar varias cosas. Que la letalidad
sea mayor del 1%, que el serotipado subestime los españoles
infectados...
14. En España con el 60% de la población infectada tendría 28.200.000 millones seropositivos, es decir, personas que ya habrían pasado la enfermedad. De estos, el 1% de muertos sería 282.000 personas. Si se infectan el 80% de los españoles, se habrían infectado 37.600.000 y la tasa de letalidad es del 1% morirían 376.000 personas.
15. En el extremo 3' de la secuencia del virus tiene una cola de poliA. Como el virus es ARN, cuando entra en el citoplasma de la célula humana tiene que tener la cola de poliA para protegerse de las ARNasas y darle tiempo a replicarse.
16. a) Los virus desnudan sus ácidos nucleicos. El problema que esto presenta es que se pueden dar encapsidaciones inespecíficas entre distintas especies de virus. b) Los seres unicelulares cuando se dividen no dejan cadaver, mientras que los seres pluricelulares, sus células se dividen en dos grupos: sexuales y las somáticas. Las sexuales tienen la oportunidad de pasar a la siguiente generación, mientras que las somáticas son solo un soporte para mantener las células sexuales de una generación a la siguiente.
17. Cuando R0 es menor que 1 la infección muere tras un largo período. Pero si R0 es mayor que 1 la infección puede llegar a propagarse ampliamente entre una población.
18. Vamos a generar una tabla de valores para luego generar la ecuación que refleje este crecimiento exponencial
19. La bacteria A en 360 minutos se divide 360/50= 7.2 veces se divide. 2 elevado a 7.2 = 147 bacterias
La bacteria B en 360 minutos se divide 360/25= 14.4 veces se divide. 2 elevado a 14.4 = 21618
Ratio (es un cociente) 21618/147 = 147 veces más bacterias B que A en 6 horas.
20. Solución pincha aquí (video inside)
21. Solución aquí
Crecimiento exponencial de las bacterias
15. En el extremo 3' de la secuencia del virus tiene una cola de poliA. Como el virus es ARN, cuando entra en el citoplasma de la célula humana tiene que tener la cola de poliA para protegerse de las ARNasas y darle tiempo a replicarse.
16. a) Los virus desnudan sus ácidos nucleicos. El problema que esto presenta es que se pueden dar encapsidaciones inespecíficas entre distintas especies de virus. b) Los seres unicelulares cuando se dividen no dejan cadaver, mientras que los seres pluricelulares, sus células se dividen en dos grupos: sexuales y las somáticas. Las sexuales tienen la oportunidad de pasar a la siguiente generación, mientras que las somáticas son solo un soporte para mantener las células sexuales de una generación a la siguiente.
17. Cuando R0 es menor que 1 la infección muere tras un largo período. Pero si R0 es mayor que 1 la infección puede llegar a propagarse ampliamente entre una población.
18. Vamos a generar una tabla de valores para luego generar la ecuación que refleje este crecimiento exponencial
#bacterias(t) = bacterias iniciales x (divisiones por hora)tiempo
en horas
a) 9.450.000.000 = 106 individuos x 2tiempo en horas
9450 = 2tiempo en horas. Para resolver log2
(9450) = ln (9450) / ln (2) = 13.2061 horas. Es decir
213,2061 = 9450, como partíamos de 106,
entoncen tendríamos 9450 x 106
b) bacterias(t) = bacterias iniciales x (divisiones por hora)tiempo
en horas. Bacterias(t) = 106 x
312. Bacterias(t) = 5,31441 x 1011
c) bacteriasB(t) = 1200000 x 3t. Al cabo de 4 hr:
bacteriasB(t) = 97.200.000
las
bacterias iniciales bacteriasA(t) = 1000.000 x 2t. Al cabo
de 4 hr: bacteriasA(t) = 16.000.000
19. La bacteria A en 360 minutos se divide 360/50= 7.2 veces se divide. 2 elevado a 7.2 = 147 bacterias
La bacteria B en 360 minutos se divide 360/25= 14.4 veces se divide. 2 elevado a 14.4 = 21618
Ratio (es un cociente) 21618/147 = 147 veces más bacterias B que A en 6 horas.
20. Solución pincha aquí (video inside)
21. Solución aquí
Crecimiento exponencial de las bacterias
| Para saber más |
domingo, 23 de septiembre de 2018
¿Son útiles los probióticos en todas las personas?
Una amiga muy querida se ha infectado comiendo hornado en el Mercado de Iñaquito. En el mercado, el cerdo casi entero está encima de unas bandejas que se recalientan todos los días. Cuando el mercado cierra el hornado se cubre y al día siguiente se vuelve a recalentar. Así, hasta que se acaba el hornado. Es fácil intoxicarse comiendo el hornado, especialmente cuando te dan hornado de los bordes de la bandeja, en esa parte la comida no alcanza los mismos grados de temperatura que la comida que está en el centro, debajo de los fogones.
Los síntomas de diarrea y malestar aparecieron 24 horas después de comer el hornado. Las tres personas que comieron hornado tuvieron los mismos síntomas. El médico le recetó un analgésico, un antiemético para que evitar las nauseas, sales para rehidratarse y un probiótico, Bacillus clausii.
¿Són útiles los probióticos? ya había escrito una entrada sobre lo inútiles que eran los yogures probióticos. Ahora, un trabajo científico publicado por el laboratorio de Eran Elinav en el Instituto Weizmann de Tel Aviv, Israel, resuelve la pregunta: los probióticos no son útiles, por mucho que la industria trate de convencernos de lo contrario.
El experimento es el típico experimento de control positivo y control negativo. ¿Qué pasa cuando nuestros intestinos carecen de bacterias después de un tratamiento antibiótico? lo que los investigadores comprobaron fue que si no tomabas probióticos la flora intestinal se regeneraba y más o menos volvía a ser la misma que antes del tratamiento antibiótico. Si después del tratamiento antibiótico se tomaban probióticos la flora intestinal ya no volvía a ser la misma porque los probióticos eran ahora una parte importante de la flora intestinal del paciente. El control negativo fue un autotransplante de heces, esto es, guardar tus heces antes del tratamiento antibiótico y después de los antibióticos inyectar las heces en el intestino.
A mi siempre me dijeron que después de un tratamiento antibiótico era bueno tomar un probiótico para regenerar la flora intestinal. Cuando mis conocidos tomaban antibióticos, con tono de sapiencia, les recomendaba un probiótico. Ahora resulta que no, que es mejor dejar que la flora se regenere por si misma si es que queremos tener la misma flora que teníamos antes.
Conclusión: con respecto a la ciencia deberíamos de marcar unas escalas, lo mismo que la escala Richter de terremotos. En medicina, la escala más alta de importancia son por ejemplo las vacunas. Las vacunas y la higiene han demostrado su eficacia y por tanto se les debe de dar la importancia que tienen. Los estudios sobre las grasas, siempre controvertidos, deberían estar en la escala más baja.
La ciencia, como método de conocimiento, en medicina funciona bien cuando las variables son pocas. Las vacunas son un ejemplo, te vacunas y ya no te contagias de ese patógeno. Cuando se quiere hacer ciencia debemos tener todas las variables controladas. Los estudios en grasas son más controvertidos porque las grasas intervienen en muuuuchos procesos.Por ese motivo, en una década te hablan del colesterol bueno y el colesterol malo y diez años después te dicen que todo eso son chorradas.
Si, a todo esto, juntamos que la industria nos "educa" constantemente y en todas partes a través de la publicidad, haciéndonos creer que por haber leído un publireportaje ya sabemos de lo que hablamos, tenemos como resultado, una población que siente que su opinión cuenta.
Ah, y por supuesto, en cuestiones de medicina nunca hagáis caso a un biólogo, como yo, porque a nosotros solo nos interesa que la selección natural elimine de la población a los portadores de genes estúpidos.
https://www.newscientist.com/article/2178860-probiotics-are-mostly-useless-and-can-actually-hurt-you/#.W5KZnmAx_FU.twitter
https://www.genomeweb.com/genetic-research/gut-microbiome-studies-question-influence-probiotics?utm_source=addthis_shares#.W5KuOxgnY8p
https://www.theguardian.com/science/2018/sep/06/probiotics-not-as-beneficial-for-gut-health-as-previously-thought?CMP=share_btn_tw
Los síntomas de diarrea y malestar aparecieron 24 horas después de comer el hornado. Las tres personas que comieron hornado tuvieron los mismos síntomas. El médico le recetó un analgésico, un antiemético para que evitar las nauseas, sales para rehidratarse y un probiótico, Bacillus clausii.
¿Són útiles los probióticos? ya había escrito una entrada sobre lo inútiles que eran los yogures probióticos. Ahora, un trabajo científico publicado por el laboratorio de Eran Elinav en el Instituto Weizmann de Tel Aviv, Israel, resuelve la pregunta: los probióticos no son útiles, por mucho que la industria trate de convencernos de lo contrario.
El experimento es el típico experimento de control positivo y control negativo. ¿Qué pasa cuando nuestros intestinos carecen de bacterias después de un tratamiento antibiótico? lo que los investigadores comprobaron fue que si no tomabas probióticos la flora intestinal se regeneraba y más o menos volvía a ser la misma que antes del tratamiento antibiótico. Si después del tratamiento antibiótico se tomaban probióticos la flora intestinal ya no volvía a ser la misma porque los probióticos eran ahora una parte importante de la flora intestinal del paciente. El control negativo fue un autotransplante de heces, esto es, guardar tus heces antes del tratamiento antibiótico y después de los antibióticos inyectar las heces en el intestino.
A mi siempre me dijeron que después de un tratamiento antibiótico era bueno tomar un probiótico para regenerar la flora intestinal. Cuando mis conocidos tomaban antibióticos, con tono de sapiencia, les recomendaba un probiótico. Ahora resulta que no, que es mejor dejar que la flora se regenere por si misma si es que queremos tener la misma flora que teníamos antes.
Conclusión: con respecto a la ciencia deberíamos de marcar unas escalas, lo mismo que la escala Richter de terremotos. En medicina, la escala más alta de importancia son por ejemplo las vacunas. Las vacunas y la higiene han demostrado su eficacia y por tanto se les debe de dar la importancia que tienen. Los estudios sobre las grasas, siempre controvertidos, deberían estar en la escala más baja.
La ciencia, como método de conocimiento, en medicina funciona bien cuando las variables son pocas. Las vacunas son un ejemplo, te vacunas y ya no te contagias de ese patógeno. Cuando se quiere hacer ciencia debemos tener todas las variables controladas. Los estudios en grasas son más controvertidos porque las grasas intervienen en muuuuchos procesos.Por ese motivo, en una década te hablan del colesterol bueno y el colesterol malo y diez años después te dicen que todo eso son chorradas.
Si, a todo esto, juntamos que la industria nos "educa" constantemente y en todas partes a través de la publicidad, haciéndonos creer que por haber leído un publireportaje ya sabemos de lo que hablamos, tenemos como resultado, una población que siente que su opinión cuenta.
Extracto de la película Idiocracia.
No, queridos lectores, por mucho que la publicidad, o los blogs de internet, te hagan creer que sabes, tu opinión es solo eso una opinión. Un médico puede hacer un diagnóstico después de examinarte y haberte hecho una serie de pruebas. Sólo un médico está capacitado para hacer un diagnóstico. Lo que diga la publicidad de probióticos, de electrolitos, de moléculas quemagrasa es solo un intento de venderte su producto.Ah, y por supuesto, en cuestiones de medicina nunca hagáis caso a un biólogo, como yo, porque a nosotros solo nos interesa que la selección natural elimine de la población a los portadores de genes estúpidos.
https://www.newscientist.com/article/2178860-probiotics-are-mostly-useless-and-can-actually-hurt-you/#.W5KZnmAx_FU.twitter
https://www.genomeweb.com/genetic-research/gut-microbiome-studies-question-influence-probiotics?utm_source=addthis_shares#.W5KuOxgnY8p
https://www.theguardian.com/science/2018/sep/06/probiotics-not-as-beneficial-for-gut-health-as-previously-thought?CMP=share_btn_tw




























