El hecho de que todos los seres vivos compartamos el mismo lenguaje genético nos permite hacer copia-corta-pega de unos genes e introducirlos de una especie a otra. Por ejemplo, podemos copiar por PCR un gen humano como puede ser la insulina e introducirlo en una bacteria para que ésta produzca la proteína humana.
Experimento explicado en video: Clonación de un gen in silico: gen gyrA
Para este ejercicio vamos a amplificar el gen gyrA de Streptococcus pneumoniae e introducirlo en el plásmido pUC19.
Copiamos el fragmento de ADN en FASTA
Entramos en ORF-Finder y pegamos el documento FASTA. De esta manera encontramos el ORF correspondiente al gen gyrA de S. pneumoniae
Entramos en ORF-Finder y pegamos el documento FASTA. De esta manera encontramos el ORF correspondiente al gen gyrA de S. pneumoniae
Copiamos el ORF1 que tiene una longitud de 2508 nt. A partir de ahora, el ORF1 correspondiente al gyrA estará resaltado en amarillo albero.
Si introducimos este ORF1 en la página Neb Cutter podremos ver qué dianas de restricción tiene este ORF1 en su secuencia
Hacemos clic en submit y este es el resultado de las dianas de restricción que existen dentro de la secuencia del Orf de gyrA.
A la derecha de esta ventana se ve un recuadro gris. Es la lista de las enzimas que no tienen diana en esa secuencia (0 cutters). Las enzimas que están en esta lista son las que nos interesan. Nos interesa diseñar los primers con una diana para una enzima que no corte el interior del orf que queremos clonar. Por ejemplo, la enzima BamHI. Vamos a elegir esta última. Esta enzima no tiene diana en el interior del orf de gyrA.
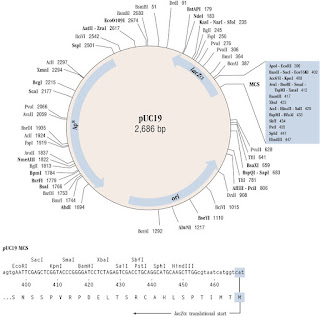
Los plásmidos tienen una región llamada polylinker que concentra una única diana para distintas enzimas de restricción
Asimismo, buscamos un plásmido que tenga un solo sitio de corte para la enzima que vamos a utilizar para cortar tanto nuestro fragmento amplificado por PCR como nuestro plásmido pUC19
En este plásmido, pUC19, archivo FASTA pUC19, solo hay una diana para BamHI. Este plásmido tiene un polylinker, que es una región de su ADN que concentra muchas dianas de restricción. Polylinker de pUC19 (en mayúsculas, en rojo la diana de BamHI):
5´agtgGAATTCGAGCTCGGTACCCGGGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTTcgtaa
Digestión pUC19 con BamHI:
El plásmido pUC19 tiene 2686 nt. Con la secuencia FASTA comprobamos que solo tiene una diana para la enzima BamHI en la applicación NEB Cutter.
5´tgGAATTCGAGCTCGGTACCCGGGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTTcg 3´
3´acCTTAAGCTCGAGCCATGGGCCCCTAGGAGATCTCAGCTGGACGTCCGTACGTTCGAAgc 5´
5´tgGAATTCGAGCTCGGTACCCGGG 3´
3´acCTTAAGCTCGAGCCATGGGCCCCTAG 5´
5´ GATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTTcg 3´
3´ GAGATCTCAGCTGGACGTCCGTACGTTCGAAgc 5´
>lcl|ORF1 CDS
5´ATGTTGTTACTTACTAATTG…………AAACAGAAGGTGAAGCATAA 3´
3´TACAACAATGAATGATTAAC…………TTTGTCTTCCACTTCGTATT 5´
Los puntos suspensivos corresponden a la secuencia del ORF que va entre los extremos del principio y del final. Para lo que es el diseño de los primers no es necesario escribirla y por eso se sustituye con puntos suspensivos.
5´ATGTTGTTACTTACTAATTG…………AAACAGAAGGTGAAGCATAA 3´
3´TTTGTCTTCCACTTCGTATT 5´
Primer EFM1
Primer EFM2
5´ATGTTGTTACTTACTAATTG 3´
3´TACAACAATGAATGATTAAC…………TTTGTCTTCCACTTCGTATT 5´
5´ATGTTGTTACTTACTAATTG…………AAACAGAAGGTGAAGCATAA 3´
3´TTTGTCTTCCACTTCGTATT 5´
Primer EFM1
Primer EFM2
5´ATGTTGTTACTTACTAATTG 3´
3´TACAACAATGAATGATTAAC…………TTTGTCTTCCACTTCGTATT 5´
Los primers EFM1 y EFM2 nos permitirían amplificar este gen. Pero queremos hacer algo más que amplificarlo. Queremos introducirlo en un plásmido para luego expresar ese gen en una bacteria.
Para ello tenemos que amplificar este ORF con dianas para una enzima de restricción que no tenga una diana en la secuencia del ORF de gyrA y que esta enzima de restricción solo tenga una diana en el plásmido en donde queremos clonar el gen. Por ejemplo, la diana para la enzima BamHI:
5´GGATCC 3´ 5´G GATCC 3´
Producto PCR con primers + diana para BamHI:
5´GCGCGGATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAAGCATAAGGATCCGCGC 3´
3´CGCGCCTAGGTACAACAATGAATGATTAAC…TTTGTCTTCCACTTCGTATTCCTAGGCGCG 5´
Ligación pUC19 + fragmento amplificado por PCR:
5´tgGAATTCGAGCTCGGTACCCGGGGATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAA
3´acCTTAAGCTCGAGCCATGGGCCCCTAGGTACAACAATGAATGATTAAC…TTTGTCTTCCACTT
GCATAAGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTTcg 3´
5´GGATCC 3´ 5´G GATCC 3´
3´CCTAGG 5´ 3´CCTAG G 5´
Diseñamos primers que incorporen la diana BamHI
Primer EFM1+BamHI: 5´ GCGCGGATCCTTATGCTTCACCTTCTGTTT 3´
Primer EFM2+BamHI: 5´ GCGCGGATCCATGTTGTTACTTACTAATTG 3´
Primer EFM1+BamHI: 5´ GCGCGGATCCTTATGCTTCACCTTCTGTTT 3´
Primer EFM2+BamHI: 5´ GCGCGGATCCATGTTGTTACTTACTAATTG 3´
A los primers, en sus extremos 5´se les añaden, además de las dianas para la enima BamHI, cuatro nucleóticos GCGC. El objeto de añadir al extremo de la cadena estos 4 nucleótidos es para que la diana GGATCC tenga la forma correcta para que la enzima se pueda unir y digerir correctamente. Si no estuviesen estos cuatro nucleótidos la cadena en sus extremos tiende a no estar enrrollada en una hélice tipo B, es decir, con 10.4 pares de bases por vuelta. Por ponerlo en palabras comunes, la cadena de ADN en su extremo está un poco "deshilachada".
Al unirse los nuevos primers van a adicionar dianas de restricción en los extremos:
5´ ATGTTGTTACTTACTAATTG…………AAACAGAAGGTGAAGCATAA 3´
3´ TTTGTCTTCCACTTCGTATTCCTAGGGCGC 5´
Primer + BamHI EFM1
Primer + BamHI EFM2
5´GCGCGGATCCATGTTGTTACTTACTAATTG 3´
3´ TACAACAATGAATGATTAAC…………TTTGTCTTCCACTTCGTATT 5´
5´GCGCGGATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAAGCATAAGGATCCGCGC 3´
3´CGCGCCTAGGTACAACAATGAATGATTAAC…TTTGTCTTCCACTTCGTATTCCTAGGCGCG 5´
Ahora este fragmento tiene 2508 nt + 10 nt extras por la izquierda y 10 nt extras por la derecha. En total 2528 nt
Producto digestión BamHI:
5´ GATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAAGCATAAG 3´
3´ GTACAACAATGAATGATTAAC…TTTGTCTTCCACTTCGTATTCCTAG 5´
Producto digestión BamHI:
5´ GATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAAGCATAAG 3´
3´ GTACAACAATGAATGATTAAC…TTTGTCTTCCACTTCGTATTCCTAG 5´
Ahora el fragmento digerido tiene 2518 nt
Ligación pUC19 + fragmento amplificado por PCR:
5´tgGAATTCGAGCTCGGTACCCGGGGATCCATGTTGTTACTTACTAATTG…AAACAGAAGGTGAA
3´acCTTAAGCTCGAGCCATGGGCCCCTAGGTACAACAATGAATGATTAAC…TTTGTCTTCCACTT
GCATAAGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTTcg 3´
CGTATTCCTAGGAGATCTCAGCTGGACGTCCGTACGTTCGAAgc 5´
¿Cuántos nucleótidos Ligación pUC19 + fragmento amplificado por PCR?
El ORF1 que tiene una longitud de 2518 nt. El plásmido pUC19 tiene 2686 nt entonces el resultado la ligación tiene 5204 nt






No hay comentarios:
Publicar un comentario
Cada vez que lees un artículo y no dejas un comentario, alguien mata a un gatito en alguna parte del mundo...